
Gaussian 16 es lo último en la serie de programas gaussianos. Proporciona capacidades de vanguardia para el modelado de estructuras electrónicas. Gaussian 16 tiene licencia para una amplia variedad de sistemas informáticos. Todas las versiones de Gaussian 16 contienen todas las funciones científicas / de modelado, y ninguna impone ninguna limitación artificial en los cálculos que no sean sus recursos informáticos y paciencia.
Continuando con la tradición de casi 40 años de la serie de programas de estructura electrónica de Gauss, Gaussian 16 ofrece nuevos métodos y capacidades que le permiten estudiar sistemas moleculares cada vez más grandes y áreas adicionales de química. GaussView 6 ofrece un amplio conjunto de capacidades de construcción y visualización. Destacamos algunas de las características más importantes en esta página.
La GFP es una proteína que emite un color verde brillante cuando se expone a la luz en el rango de azul a ultravioleta. El cromóforo se muestra en el recuadro a continuación. La molécula se aisló por primera vez en la especie de medusa Aequorea victoria , que es nativa de la costa noroeste del Pacífico de América del Norte. Desde entonces, se ha estudiado ampliamente y se han diseñado variantes de la molécula con propiedades de fluorescencia mejoradas.
GFP consiste en un cromóforo dentro de una cadena de proteínas compuesta por 238 aminoácidos. El cromóforo aislado no es fluorescente, por lo que moldearlo en su entorno de proteínas es esencial. El ciclo de fluorescencia de GFP implica una excitación inicial a su primer estado excitado, una reacción de transferencia de protones en la superficie de energía potencial S 1 y, finalmente, una relajación al estado fundamental.
Las siguientes características de Gaussian 16 y GaussView 6 son útiles para modelar la fluorescencia en este compuesto:
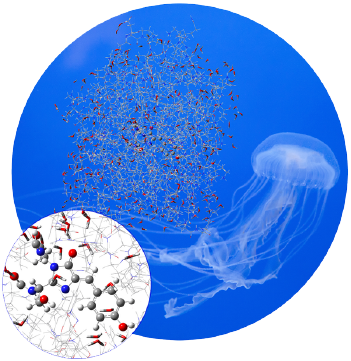
GaussView puede abrir directamente archivos desde el banco de datos de proteínas (archivos PDB). Puede agregar hidrógenos a la estructura recuperada cuando se importa o en un momento posterior. También puede ver, manipular y modificar la estructura utilizando la cadena de PDB, el tipo y / o número de residuo, y otra información de subestructura de PDB presente en el archivo original. Los residuos y otra información pueden ser retenidos durante la edición de la molécula y la ejecución del trabajo.
La molécula se puede modelar mediante cálculos de MO: MM utilizando la función ONIOM de Gaussian:


Mar
12
2026

Mar
24
2026
 LATAM
LATAM

Abr
10
2026

Abr
16
2026

Abr
17
2026

May
29
2026

